Bio.PDB 是 Biopython 的一個模組,專注於處理生物巨分子的晶體結構。本文檔提供了 Bio.PDB 的相當完整的概述。
Bio.PDB 會自動作為 Biopython 的一部分安裝。Biopython 可以從 http://www.biopython.org 取得。它可以在許多平台上執行 (Linux/Unix、Windows、Mac 等)。
Bio.PDB 被用於建構 DISEMBL,這是一個預測蛋白質中無序區域的網路伺服器,以及 COLUMBA,一個提供註釋蛋白質結構的網站(不再可用?)。Bio.PDB 也被用於在 PDB 中執行蛋白質結構之間活性位點相似性的大規模搜索(請參閱 Proteins 51: 96–108, 2003),並開發一種新的演算法來識別線性二級結構元素(請參閱 BMC Bioinformatics 6: 202, 2005)。
從功能和資訊的需求來看,Bio.PDB 也被幾家大型製藥公司 (LPC) 使用 :-)。
有的,如果您使用了 Bio.PDB,我會很感激您在出版物中引用它。參考文獻是
Hamelryck, T., Manderick, B. (2003) PDB parser and structure class implemented in Python. Bioinformatics 19: 2308–2310
這篇文章可以透過 生物資訊學期刊網站 免費下載。我歡迎您發送電子郵件告訴我您使用 Bio.PDB 的用途。功能請求也歡迎。
實際上相當好。Bio.PDB 已經在來自 PDB 的近 5500 個結構上進行了廣泛的測試 - 所有結構似乎都得到了正確的解析。更多詳細資訊可以在 Bio.PDB 生物資訊學文章中找到。Bio.PDB 已經/正在許多研究專案中作為可靠的工具使用。事實上,我幾乎每天都將 Bio.PDB 用於研究目的,並繼續致力於改進它並添加新功能。
PDBParser 的效能在約 800 個結構(每個結構都屬於一個獨特的 SCOP 超家族)上進行了測試。這需要大約 20 分鐘,或平均每個結構 1.5 秒。解析大型核醣體亞基 (1FKK) 的結構,其中包含約 64000 個原子,在 1000 MHz 的 PC 上需要 10 秒。簡而言之:對於許多應用來說,它已經足夠快了。
Bio.PDB 可能正是您想要的,但也可能不是。如果您對 PDB 標頭的資料探勘感興趣,您可能需要考慮其他地方,因為這方面的支援有限。如果您正在尋找一個強大、完整的資料結構來存取原子資料,那麼 Bio.PDB 可能適合您。
這很簡單
from Bio.PDB import *
不是直接支援,主要是因為已經有很多基於 Python 或具有 Python 感知的解決方案,它們有可能與 Bio.PDB 一起使用。我選擇的是 Pymol,順便說一句(我已經成功地將它與 Bio.PDB 一起使用,而且 Bio.PDB 中可能很快/有一天會出現特定的 PyMol 模組)。基於 Python 或具有 Python 感知的分子圖形解決方案包括
再寫一個分子圖形應用程式會很瘋狂(實際上已經做過了 :-)。
但是,您可以使用 nglview 在 Jupyter 筆記本中以互動方式檢視 Biopython 結構實體
import nglview as nv
view = nv.show_biopython(structure)
view
首先,建立一個 PDBParser 物件
parser = PDBParser()
然後,以下列方式從 PDB 檔案建立結構物件(在這種情況下,PDB 檔案名為 1FAT.pdb,PHA-L 是使用者定義的結構名稱)
structure = parser.get_structure("PHA-L", "1FAT.pdb")
與 PDB 檔案的情況類似,首先建立一個 MMCIFParser 物件
parser = MMCIFParser()
然後使用此解析器從 mmCIF 檔案建立結構物件
structure = parser.get_structure("PHA-L", "1FAT.cif")
目前尚不支援,但我絕對計畫在未來支援(這不是很多工作)。如果您需要此功能,請與我聯繫,這可能會鼓勵我 :-)。
沒問題。您可以建立一個 Python 字典,將 mmCIF 檔案中的所有 mmCIF 標籤對應到它們的值。如果有多個值(例如標籤 _atom_site.Cartn_y,它保存所有原子的y座標),則該標籤會對應到一個值列表。字典是從 mmCIF 檔案中建立的,如下所示
from Bio.PDB.MMCIF2Dict import MMCIF2Dict
mmcif_dict = MMCIF2Dict("1FAT.cif")
範例:從 mmCIF 檔案取得溶劑含量
sc = mmcif_dict["_exptl_crystal.density_percent_sol"]
範例:取得所有原子的 y 座標列表
y_list = mmcif_dict["_atom_site.Cartn_y"]
感謝 Christian Rother,您可以存取 PDB 標頭中的一些資訊。但請注意,許多 PDB 檔案包含具有不完整或錯誤資訊的標頭。許多錯誤已在等效的 mmCIF 檔案中修正。因此,如果您對標頭資訊感興趣,最好使用上述 MMCIF2Dict 工具從 mmCIF 檔案中提取資訊,而不是解析 PDB 標頭。
現在已經澄清了,讓我們回到解析 PDB 標頭。結構物件有一個名為 header 的屬性,它是一個 Python 字典,將標頭記錄對應到它們的值。
範例
resolution = structure.header["resolution"]
keywords = structure.header["keywords"]
可用的索引鍵包括 name、head、deposition_date、release_date、structure_method、resolution、structure_reference(對應到參考文獻列表)、journal_reference、author 和 compound(對應到包含有關結晶化合物的各種資訊的字典)。
也可以在不建立 Structure 物件的情況下,直接從 PDB 檔案建立字典
handle = open(filename, "r")
header_dict = parse_pdb_header(handle)
handle.close()
當然。許多 PDB 解析器假設只有一個模型,這使得它們對於 NMR 結構幾乎毫無用處。Structure 物件的設計使其可以輕鬆處理具有多個模型的 PDB 檔案(請參閱 結構物件 章節)。
可以使用 PDBList 物件,使用 retrieve_pdb_file 方法來完成。此方法的引數是結構的 PDB 識別符號。
pdbl = PDBList()
pdbl.retrieve_pdb_file("1FAT")
PDBList 類別也可以作為命令列工具使用
python PDBList.py 1fat
下載的檔案將被命名為 pdb1fat.ent 並儲存在目前的工作目錄中。請注意,retrieve_pdb_file 方法還有一個可選引數 pdir,用於指定儲存下載的 PDB 檔案的特定目錄。
retrieve_pdb_file 方法還有一些選項,用於指定下載使用的壓縮格式,以及用於本地解壓縮的程式(預設為 .Z 格式和 gunzip)。此外,可以在建立 PDBList 物件時指定 PDB ftp 站點。預設情況下,使用 全球蛋白質資料庫 的 ftp 伺服器。有關更多詳細資訊,請參閱 API 文件。再次感謝 Kristian Rother 捐贈此模組。
以下命令會將所有 PDB 檔案儲存在 /data/pdb 目錄中
python PDBList.py all /data/pdb
python PDBList.py all /data/pdb -d
此 API 方法稱為 download_entire_pdb。新增 -d 選項會將所有檔案儲存在同一目錄中。否則,它們會根據其 PDB ID 分類到 PDB 樣式的子目錄中。根據流量的不同,完整下載需要 2-4 天。
也可以使用 PDBList 物件來完成。只需建立一個 PDBList 物件(指定 PDB 的本地副本所在的目錄)並呼叫 update_pdb 方法即可
pl = PDBList(pdb="/data/pdb")
pl.update_pdb()
當然,可以將此操作設定為每週的 cronjob,以便自動使本地副本保持最新。也可以指定 PDB ftp 站點(請參閱 API 文件)。
PDBList 還有一些額外的方法可以使用。get_all_obsolete 方法可以用來取得所有過時的 PDB 條目的列表。changed_this_week 方法可以用來取得本週新增、修改或過時的條目。關於 PDBList 的更多可能性,請參閱 API 文件。
眾所周知,許多 PDB 檔案都包含語義錯誤(我指的不是結構本身,而是它們在 PDB 檔案中的表示)。Bio.PDB 嘗試以兩種方式處理這個問題。PDBParser 物件可以兩種方式運作:嚴格模式和寬鬆模式(現在是預設)。嚴格模式過去是預設模式,但人們似乎認為 Bio.PDB 因為一個錯誤而「崩潰」(哈!),所以我更改了它。如果您遇到真正的錯誤,請立即告訴我!
範例
# Permissive parser
parser = PDBParser(PERMISSIVE=1)
parser = PDBParser() # The same (default)
# Strict parser
strict_parser = PDBParser(PERMISSIVE=0)
在寬鬆模式(預設)下,明顯包含錯誤的 PDB 檔案會被「修正」(即,某些殘基或原子會被忽略)。這些錯誤包括:
這些錯誤表示 PDB 檔案中存在真正的問題(詳細資訊請參閱 Bioinformatics 文章)。在嚴格模式下,包含錯誤的 PDB 檔案會導致例外發生。這對於找出 PDB 檔案中的錯誤很有用。
然而,有些錯誤會自動修正。通常,每個無序原子都應該有一個非空白的 altloc 識別符。然而,有許多結構不遵循這個慣例,並且對於同一個原子的兩個無序位置,同時具有空白和非空白的識別符。這會自動以正確的方式解釋。
有時,一個結構包含屬於鏈 A 的殘基列表,接著是屬於鏈 B 的殘基,然後又是屬於鏈 A 的殘基,也就是說,這些鏈是「斷裂的」。這也會被正確解釋。
使用 PDBIO 類別來實現。當然,也很容易寫出結構的特定部分。
範例:儲存一個結構
io = PDBIO()
io.set_structure(s)
io.save("out.pdb")
如果您想寫出結構的一部分,請使用 Select 類別(也在 PDBIO 中)。Select 有四個方法:
accept_model(model)
accept_chain(chain)
accept_residue(residue)
accept_atom(atom)
預設情況下,每個方法都會回傳 1(表示模型/鏈/殘基/原子包含在輸出中)。透過繼承 Select 並在適當時回傳 0,您可以從輸出中排除模型、鏈等等。也許很麻煩,但非常強大。以下程式碼只會寫出甘胺酸殘基:
class GlySelect(Select):
def accept_residue(self, residue):
if residue.get_name() == "GLY":
return 1
else:
return 0
io = PDBIO()
io.set_structure(s)
io.save("gly_only.pdb", GlySelect())
如果這對您來說太複雜了,Dice 模組包含一個方便的 extract 函式,它可以寫出鏈中開始和結束殘基之間的所有殘基。
不,而且我也不打算很快(或永遠)添加該功能(我根本不需要它,而且這需要大量工作,而且從來沒有人要求過)。想要添加這個功能的人可以聯絡我。
Structure 物件遵循所謂的 SMCRA(結構/模型/鏈/殘基/原子)架構:
這是許多結構生物學家/生物資訊學家思考結構的方式,並提供了一種簡單但有效的方式來處理結構。其他額外的東西基本上是在需要時添加的。下面圖中顯示了 Structure 物件的 UML 圖(暫時忽略 Disordered 類別)。
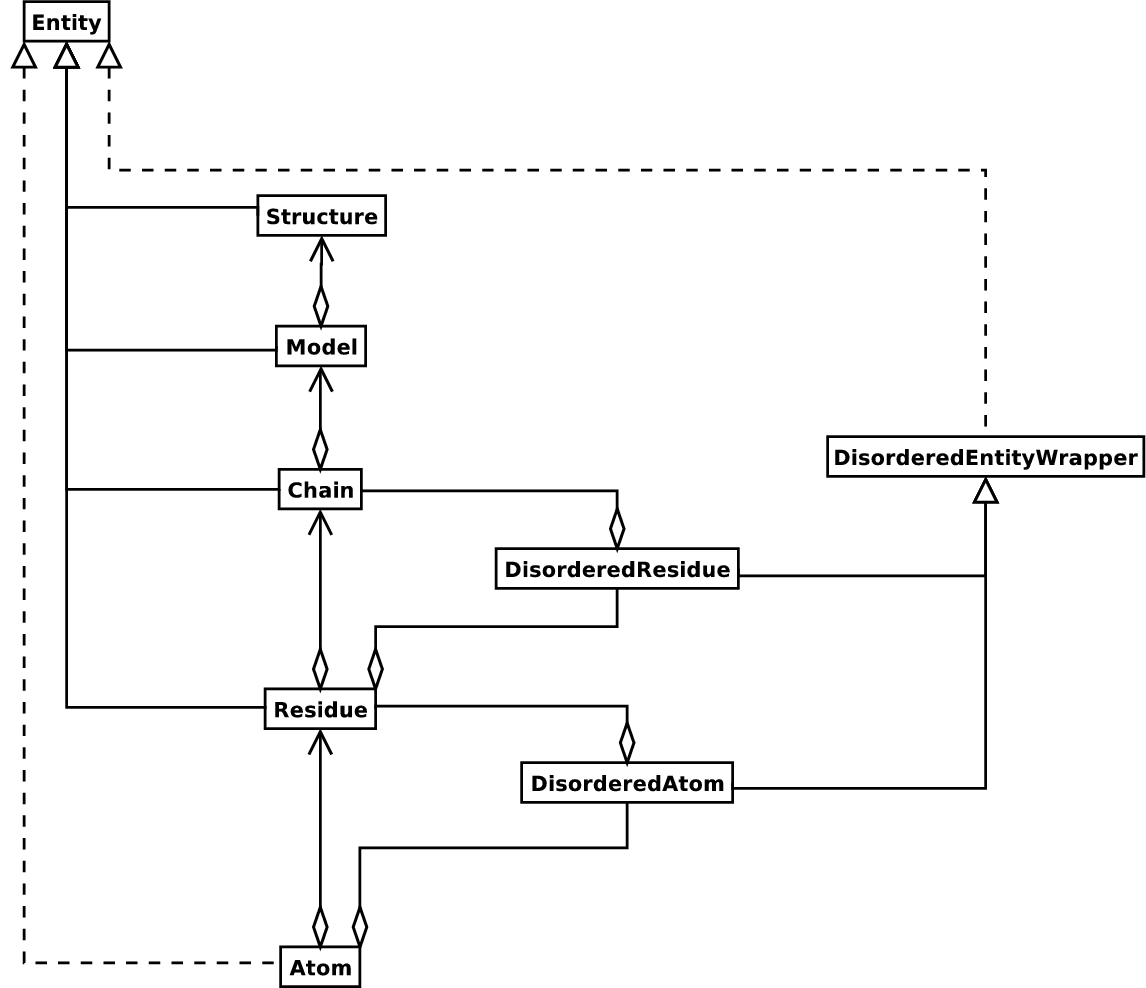
結構物件的 SMCRA 架構圖。
帶有菱形的全線表示聚合,帶有箭頭的全線表示引用,帶有三角形的全線表示繼承,帶有三角形的虛線表示介面實現。
以下程式碼會迭代瀏覽結構的所有原子:
p = PDBParser()
structure = p.get_structure("X", "pdb1fat.ent")
for model in structure:
for chain in model:
for residue in chain:
for atom in residue:
print(atom)
還有一些捷徑:
# Iterate over all atoms in a structure
for atom in structure.get_atoms():
print(atom)
# Iterate over all residues in a model
for residue in model.get_residues():
print(residue)
結構、模型、鏈、殘基和原子在 Biopython 中稱為 實體。您始終可以從子 Entity 取得父 Entity,例如:
residue = atom.get_parent()
chain = residue.get_parent()
您也可以使用 has_id 方法來測試 Entity 是否具有特定的子實體。
您可以執行以下操作:
atoms = structure.get_atoms()
residues = structure.get_residues()
atoms = chain.get_atoms()
您也可以使用 Selection.unfold_entities 函式:
# Get all residues from a structure
res_list = Selection.unfold_entities(structure, "R")
# Get all atoms from a chain
atom_list = Selection.unfold_entities(chain, "A")
顯然,A=原子,R=殘基,C=鏈,M=模型,S=結構。您可以使用這個方法在階層中向上移動,例如,從 Atom 的列表取得(唯一的)Residue 或 Chain 父節點列表:
residue_list = Selection.unfold_entities(atom_list, "R")
chain_list = Selection.unfold_entities(atom_list, "C")
如需更多資訊,請參閱 API 文件。
Structure 中提取特定的 Atom/Residue/Chain/Model?很簡單。這裡有一些範例:
model = structure[0]
chain = model["A"]
residue = chain[100]
atom = residue["CA"]
請注意,您可以使用捷徑:
atom = structure[0]["A"][100]["CA"]
模型 ID 是一個整數,表示模型在 PDB/mmCIF 檔案中的排序。模型 ID 從 0 開始。晶體結構通常只有一個模型(id 為 0),而 NMR 檔案通常有幾個模型。
鏈 ID 在 PDB/mmCIF 檔案中指定,並且是一個單一字元(通常是字母)。
由於 PDB 格式的笨拙,這有點複雜。殘基 ID 是一個包含三個元素的元組:
'H_' 加上異質殘基的名稱(例如,葡萄糖分子的情況下為 'H_GLC'),或水分子情況下的 'W'。'A'。插入碼有時用於保留某種期望的殘基編號方案。例如,Ser 80 插入突變體(例如,插入在 Thr 80 和 Asn 81 殘基之間)的序列識別符和插入碼可能如下:Thr 80 A、Ser 80 B、Asn 81。這樣,殘基編號方案就可以與野生型結構的編號方案保持一致。因此,上述葡萄糖殘基的 id 將會是 ('H_GLC', 100, 'A')。如果異質旗標和插入碼為空白,則可以單獨使用序列識別符。
# Full id
residue = chain[(" ", 100, " ")]
# Shortcut id
residue = chain[100]
使用異質旗標的原因是,許多 PDB 檔案對氨基酸以及異質殘基或水使用相同的序列識別符,如果沒有使用異質旗標,就會產生明顯的問題。
原子 ID 只是原子名稱(例如,'CA')。實際上,原子名稱是透過移除 PDB 檔案中原子名稱的所有空格來建立的。
但是,在 PDB 檔案中,空格可以是原子名稱的一部分。通常,鈣原子被稱為 'CA..',以便將它們與 Cα 原子(稱為 '.CA.')區分開來。在移除空格會造成問題的情況下(即,同一個殘基中有兩個稱為 'CA' 的原子),則會保留空格。
這是 Bio.PDB 的強項之一。它可以處理無序原子和點突變(即,相同位置的 Gly 和 Ala 殘基)。
應該從兩個角度來看待無序:原子和殘基的角度。總體而言,我試圖封裝由無序產生的所有複雜性。如果您只想遍歷所有 Cα 原子,您不會關心某些殘基是否具有無序的側鏈。另一方面,也應該有可能在資料結構中完全表示無序。因此,無序原子或殘基儲存在特殊的物件中,這些物件的行為就像沒有無序一樣。這是透過只表示無序原子或殘基的子集來完成的。選擇哪個子集(例如,使用 Ser 殘基的兩個無序 OG 側鏈原子位置中的哪一個)可以由使用者指定。
無序原子位置由普通的 Atom 物件表示,但是所有表示相同物理原子的 Atom 物件都儲存在一個 DisorderedAtom 物件中(請參閱結構物件章節)。DisorderedAtom 物件中的每個 Atom 物件都可以使用其 altloc 指定符來唯一索引。DisorderedAtom 物件會將所有未捕獲的方法呼叫轉發到選定的 Atom 物件,預設情況下是代表具有最高佔有率的原子的物件。使用者當然可以變更選定的 Atom 物件,並使用其 altloc 指定符。這樣,就可以正確地表示原子無序,而不會增加太多的複雜性。換句話說,如果您對原子無序不感興趣,就不會受到它的干擾。
每個無序原子都有一個特徵的 altloc 識別符。您可以指定 DisorderedAtom 物件的行為應該像與特定 altloc 識別符相關聯的 Atom 物件一樣。
atom.disordered_select("A") # select altloc A atom
atom.disordered_select("B") # select altloc B atom
當無序是由於點突變造成時,就會出現特殊情況,也就是說,當晶體中存在多個多肽的點突變體時。一個例子可以在 PDB 結構 1EN2 中找到。
由於這些殘基屬於不同的殘基類型(例如,假設 Ser 60 和 Cys 60),因此它們不應該像一般情況一樣儲存在單一的 Residue 物件中。在這種情況下,每個殘基都由一個 Residue 物件表示,並且兩個 Residue 物件都儲存在單一的 DisorderedResidue 物件中(請參閱結構物件章節)。
DisorderedResidue 物件會將所有未捕獲的方法轉發到選定的 Residue 物件(預設情況下是最後添加的 Residue 物件),因此其行為就像一個普通的殘基。DisorderedResidue 物件中的每個 Residue 物件都可以透過其殘基名稱來唯一識別。在上述範例中,殘基 Ser 60 在 DisorderedResidue 物件中的 id 將會是 'SER',而殘基 Cys 60 的 id 將會是 'CYS'。使用者可以透過此 id 來選取 DisorderedResidue 物件中的活動 Residue 物件。
範例:假設一個鏈在位置 10 有一個點突變,由一個 Ser 和一個 Cys 殘基組成。請確保此鏈的位置 10 的殘基表現得像 Cys 殘基。
residue = chain[10]
residue.disordered_select("CYS")
此外,您可以使用 (Disordered)Residue 物件的 get_unpacked_list 方法,取得所有 Atom 物件的列表(即,所有 DisorderedAtom 物件都會「解包」為其個別的 Atom 物件)。
可以,某種程度上可以,但我正在等待這個功能的請求完成 :-)。
請參閱「什麼是殘基 ID?」。
是的!Bio.PDB 支援等向性和非等向性 B 因子,並且在存在非等向性 B 因子的標準差時也會處理(請參閱分析章節)。
是的,支援。請參閱分析章節。
當然,當然。每個人總是提出(大多是空談或部分實作的)資料結構,以處理所有可能的情況,並以所有可想像(和不可想像)的方式擴展。然而,平淡的真相是,使用(我的意思是真的使用!)晶體結構的人中,有 99.9% 是以模型、鏈、殘基和原子的角度思考。 Bio.PDB 的理念是提供一個相當快速、乾淨、簡單但完整的方式來存取結構資料。布丁的好壞,吃過才知道。
此外,在 Structure 類別之上建立更專業的資料結構相當容易(例如,有一個 Polypeptide 類別)。另一方面,Structure 物件是使用 Parser/Consumer 方法(分別稱為 PDBParser/MMCIFParser 和 StructureBuilder)建立的。可以透過實作專業的 StructureBuilder 類別,輕鬆重複使用 PDB/mmCIF 解析器。當然,透過編寫新的解析器,新增對新檔案格式的支援也很簡單。
Atom 物件中提取資訊?使用以下方法
a.get_name() # atom name (spaces stripped, e.g. 'CA')
a.get_id() # id (equals atom name)
a.get_coord() # atomic coordinates
a.get_vector() # atomic coordinates as Vector object
a.get_bfactor() # isotropic B factor
a.get_occupancy() # occupancy
a.get_altloc() # alternative location specifier
a.get_sigatm() # std. dev. of atomic parameters
a.get_siguij() # std. dev. of anisotropic B factor
a.get_anisou() # anisotropic B factor
a.get_fullname() # atom name (with spaces, e.g. '.CA.')
Residue 物件中提取資訊?使用以下方法
r.get_resname() # return the residue name (eg. 'GLY')
r.is_disordered() # 1 if the residue has disordered atoms
r.get_segid() # return the SEGID
r.has_id(name) # test if a residue has a certain atom
這很簡單:原子的減號運算子已重載為傳回兩個原子之間的距離。
範例
# Get some atoms
ca1 = residue1["CA"]
ca2 = residue2["CA"]
# Simply subtract the atoms to get their distance
distance = ca1 - ca2
這可以透過原子坐標的向量表示法,以及 Vector 模組中的 calc_angle 函式輕鬆完成
vector1 = atom1.get_vector()
vector2 = atom2.get_vector()
vector3 = atom3.get_vector()
angle = calc_angle(vector1, vector2, vector3)
同樣地,這可以透過原子坐標的向量表示法輕鬆完成,這次使用 Vector 模組中的 calc_dihedral 函式
vector1 = atom1.get_vector()
vector2 = atom2.get_vector()
vector3 = atom3.get_vector()
vector4 = atom4.get_vector()
angle = calc_dihedral(vector1, vector2, vector3, vector4)
使用 NeighborSearch。這在幕後使用 C 語言編碼的 KD 樹狀資料結構,因此速度相當快(請參閱 Bio.KDTree)。
Structure 物件中提取多肽?使用 PolypeptideBuilder。您可以使用產生的 Polypeptide 物件,取得序列作為 Seq 物件,或取得 Cα 原子的列表。可以使用 C-N 或 Cα-Cα 距離標準來建立多肽。
範例
# Using C-N
ppb = PPBuilder()
for pp in ppb.build_peptides(structure):
print(pp.get_sequence())
# Using CA-CA
ppb = CaPPBuilder()
for pp in ppb.build_peptides(structure):
print(pp.get_sequence())
請注意,在上述情況下,PolypeptideBuilder 只會考慮結構的模型 0。但是,也可以使用 PolypeptideBuilder 從 Model 和 Chain 物件建立 Polypeptide 物件。
首先要做的就是從結構中提取所有多肽(請參閱前一個條目)。然後可以從 Polypeptide 物件輕鬆取得每個多肽的序列。序列以 Biopython Seq 物件表示。
範例
>>> seq = polypeptide.get_sequence()
>>> print(seq)
Seq('SNVVE...')
對於此功能,您需要安裝 DSSP(並取得其授權 - 學術用途免費)。然後使用 DSSP 類別,將 Residue 物件對應到其二級結構(和可及表面積)。DSSP 代碼列於下表中。請注意,DSSP(程式以及因此的類別)無法處理多個模型!
| DSSP 代碼 | 二級結構 |
|---|---|
| H | α-螺旋 |
| B | 孤立 β-橋殘基 |
| E | 鏈 |
| G | 3-10 螺旋 |
| I | Π-螺旋 |
| T | 轉角 |
| S | 彎曲 |
| - | 其他 |
使用 DSSP 類別(另請參閱前一個條目)。但另請參閱下一個條目。
殘基深度是殘基的原子與溶劑可及表面之間的平均距離。它是溶劑可及性的一個相當新且非常強大的參數化方法。對於此功能,您需要安裝 Michel Sanner 的 MSMS 程式。然後使用 ResidueDepth 類別。這個類別的行為像一個字典,將 Residue 物件對應到相應的(殘基深度、Cα 深度)元組。Cα 深度是殘基的 Cα 原子到溶劑可及表面的距離。
範例
model = structure[0]
rd = ResidueDepth(model, pdb_file)
residue_depth, ca_depth = rd[some_residue]
您也可以存取分子表面本身(透過 get_surface 函式),其形式為帶有表面點的 NumPy 陣列。
半球暴露 (HSE) 是一種新的 2D 溶劑暴露測量方法。基本上,它計算殘基周圍沿著其側鏈方向和相反方向(在 13 Å 的半徑內)的 Cα 原子數量。儘管它很簡單,但它比許多其他溶劑暴露的測量方法更有效。一篇描述這種新型 2D 測量方法的文章已提交。
HSE 有兩種形式:HSEα 和 HSEβ。前者只使用 Cα 原子位置,而後者則使用 Cα 和 Cβ 原子位置。HSE 測量由 HSExposure 類別計算,該類別也可以計算接觸數。後者類別具有傳回字典的方法,這些字典會將 Residue 物件對應到其相應的 HSEα、HSEβ 和接觸數值。
範例
model = structure[0]
hse = HSExposure()
# Calculate HSEalpha
exp_ca = hse.calc_hs_exposure(model, option="CA3")
# Calculate HSEbeta
exp_cb = hse.calc_hs_exposure(model, option="CB")
# Calculate classical coordination number
exp_fs = hse.calc_fs_exposure(model)
# Print HSEalpha for a residue
print(exp_ca[some_residue])
首先,建立 FASTA 格式的比對檔案,然後使用 StructureAlignment 類別。這個類別也可以用於兩個以上結構的比對。
使用 is_aa(residue)。
Atom 物件使用 get_vector 方法傳回坐標的 Vector 物件表示法。Vector 實作完整的 3D 向量運算、矩陣乘法(左和右)以及一些進階的旋轉相關運算。另請參閱下一個問題。
好吧,我承認,這個範例只是為了展示 Bio.PDB 的 Vector 模組的可能性而存在的(儘管這個程式碼實際上是用在 HSExposure 模組中,其中包含參數化殘基暴露的新方法 - 出版中)。假設您想找到 Gly 殘基的 Cβ 原子位置,如果它有的話。您會怎麼做?嗯,將 Gly 殘基的 N 原子沿著 Cα-C 鍵旋轉 -120 度,大約會將其置於虛擬 Cβ 原子的位置。以下是如何使用 Vector 模組的 rotaxis 方法(可以用來建構繞著特定軸的旋轉)來完成此操作
# get atom coordinates as vectors
n = residue["N"].get_vector()
c = residue["C"].get_vector()
ca = residue["CA"].get_vector()
# center at origin
n = n - ca
c = c - ca
# find rotation matrix that rotates n -120 degrees along the ca-c vector
rot = rotaxis(-pi * 120.0 / 180.0, c)
# apply rotation to ca-n vector
cb_at_origin = n.left_multiply(rot)
# put on top of ca atom
cb = cb_at_origin + ca
這個範例顯示,有可能對原子資料執行一些相當重要的向量運算,這可能相當有用。除了所有常見的向量運算(叉積(使用 **),和點積(使用 *)運算、角度、範數等等)以及上述的 rotaxis 函式之外,Vector 模組還具有將一個向量旋轉 (rotmat) 或反射 (refmat) 到另一個向量之上的方法。
令人驚訝的是,這是使用 Superimposer 物件完成的。此物件會計算旋轉和平移矩陣,以將兩個原子列表彼此旋轉疊合,使其 RMSD(均方根偏差)最小化。當然,這兩個列表需要包含相同數量的原子。Superimposer 物件也可以將旋轉/平移應用於原子列表。旋轉和平移以元組形式儲存在 Superimposer 物件的 rotran 屬性中(請注意,旋轉是右乘!)。RMSD 儲存在 rmsd 屬性中。
Superimposer 使用的演算法來自 Matrix computations, 2nd ed. Golub, G. & Van Loan (1989),並利用奇異值分解(這在一般的 Bio.SVDSuperimposer 模組中實現)。
範例
sup = Superimposer()
# Specify the atom lists
# 'fixed' and 'moving' are lists of Atom objects
# The moving atoms will be put on the fixed atoms
sup.set_atoms(fixed, moving)
# Print rotation/translation/rmsd
print(sup.rotran)
print(sup.rms)
# Apply rotation/translation to the moving atoms
sup.apply(moving)
非常簡單。使用活性位點原子計算旋轉/平移矩陣(見上文),然後將這些矩陣應用於整個分子。
可以,使用 Atom 物件的 transform 方法,或直接使用 set_coord 方法。
請參閱主要的 Biopython 教學。
目前尚無文件。
哇!時間晚了,我累了,而且一杯極品的 Pedro Ximenez 雪利酒正在等著我。請給我發郵件,我會在早上回答你(如果幸運的話……)。
Bio.PDB 的主要作者/維護者是
Thomas Hamelryck 生物資訊學中心 哥本哈根大學分子生物學研究所 Universitetsparken 15, Bygning 10 DK-2100 København Ø 丹麥
Kristian Rother 捐贈了與 PDB 資料庫互動以及解析 PDB 表頭的程式碼。Indraneel Majumdar 發送了一些錯誤報告並協助編寫了 Polypeptide 模組。非常感謝 Brad Chapman、Jeffrey Chang、Andrew Dalke 和 Iddo Friedberg 的建議、評論、幫助和/或嚴厲批評 :-)。
可以,可以,可以!如果您有任何有用的東西可以貢獻,請發送電子郵件給 Thomas Hamelryck 或 Biopython 開發人員!永恆的名聲在等著您!