ABI 數據追蹤檔案對於檢查很有用。雖然有現成的解決方案可用,但您可能會發現自己需要編寫自己的程式碼來以程式化的方式與數據追蹤互動。Biopython 允許我們這樣做。
我將在下面展示一個互動式的 IPython 終端機工作階段,使用我自己的 Sanger 定序檔案,但它也適用於其他 AB1 檔案。
首先,我們讀取 ABI 檔案
from Bio import SeqIO
record = SeqIO.read("55-Mn-fw-EM-28.ab1", "abi")
我們最感興趣的資料位於記錄的 annotations 屬性中。
>>> list(record.annotations.keys())
dict_keys(["dye", "abif_raw", "sample_well", "run_finish", "machine_model", "run_start", "polymer"])
在 abif_raw 下面是另一個資料字典。
>>> list(record.annotations["abif_raw"].keys())
dict_keys(["DATA5", "DATA8", "RUNT1", "phAR1", ..., "DATA6"])
根據 ABI 資料規格(第 40 頁),傳統顯示的數據追蹤所需的所有資料都在 DATA9 到 DATA12 通道中。我們可以透過程式化的方式抓取這些通道。但是,沒有清楚說明哪些字母對應哪些顏色,因此也沒有說明哪個確切的通道。
from collections import defaultdict
channels = ["DATA9", "DATA10", "DATA11", "DATA12"]
trace = defaultdict(list)
for c in channels:
trace[c] = record.annotations["abif_raw"][c]
現在,可以在 matplotlib 圖形上繪製它們。
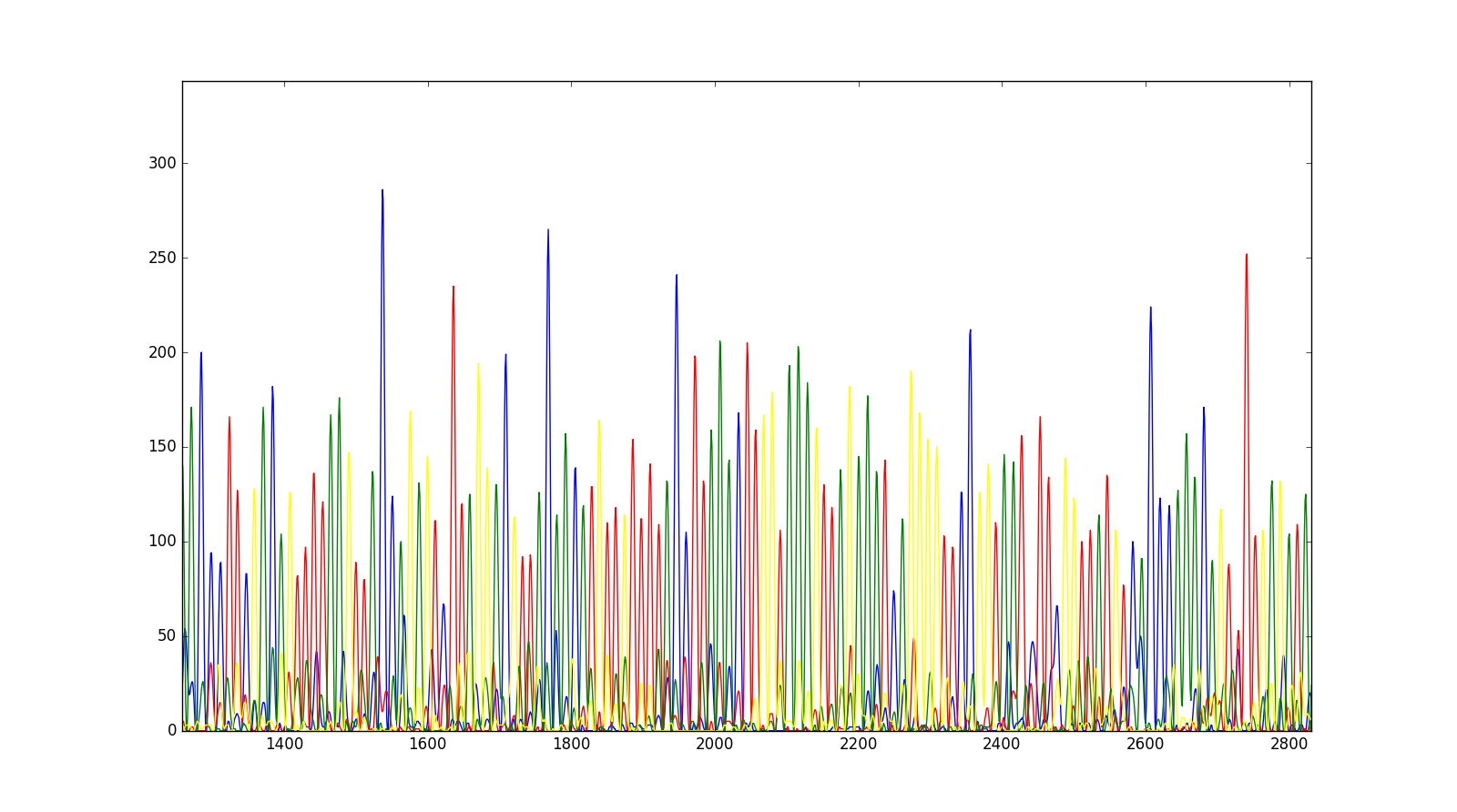
plt.plot(trace["DATA9"], color="blue")
plt.plot(trace["DATA10"], color="red")
plt.plot(trace["DATA11"], color="green")
plt.plot(trace["DATA12"], color="yellow")
plt.show()
在放大到特定區域後,我們將得到以下數據追蹤圖。
截至目前撰寫時,這個食譜並未進一步計算指標。一個特別有趣的事情是量化混合鹼基呼叫與單一鹼基呼叫的香農多樣性。
另一個需要注意的事項:資料中每個鹼基有 10 個色譜圖值。因此,這表示建議抓取每第 5 個值,以便序列數據追蹤陣列的最終長度與應該定序的位置數量相符,而不是大 10 倍。